WGATOOLS 开发与使用
由来
在我的 上一篇文章 中,我说我需要对多种全基因组比对格式进行转换,以适应我下游的分析需求。然后我发现实际上下游很多分析,应该是可以适配多种不同的格式的,还有许多个性化的分析也很常用。正好市面上似乎没有这类大整合的工具,同时我的 rust 语言熟练程度也来到了自信区😄, 所以就动手写了一个工具套件,取名为 wgatools = W hole G enome A lignment Tools
目前该工具已经开源在 github 上,欢迎大家使用 \ 提建议 \ 点赞。
安装
安装很简单,纯纯的 rust 写的,直接 cargo 安装就完事了,两种方式:
- 手动克隆 repo 编译
git clone https://github.com/wjwei-handsome/wgatools.git
cd wgatools
cargo build --release
./target/release/wgatools
- 直接指定 repo 安装
cargo install --git https://github.com/wjwei-handsome/wgatools.git
使用
和市面上绝大多数的现代工具套件一样,采用 wgatools <subcommand> <options> 的形式进行调用,如下所示:
$ wgatools
wgatools -- a cross-platform and ultrafast toolkit for Whole Genome Alignment Files manipulation
Version: 0.1.0
Authors: Wenjie Wei <wjwei9908@gmail.com>
Usage: wgatools [OPTIONS] <COMMAND>
Commands:
maf2paf Convert MAF format to PAF format [aliases: m2p]
maf2chain Convert MAF format to Chain format [aliases: m2c]
paf2maf Convert PAF format to MAF format [aliases: p2m]
paf2chain Convert PAF format to Chain format [aliases: p2c]
chain2maf Convert Chain format to MAF format [aliases: c2m]
chain2paf Convert Chain format to PAF format [aliases: c2p]
maf-index Build index for MAF file [aliases: mi]
maf-ext Extract specific region from MAF file with index [aliases: me]
call Call Variants from MAF file [aliases: c]
tview View MAF file in terminal [aliases: tv]
stat Statistics for Alignment file [aliases: st]
dotplot TEST: Plot dotplot for Alignment file [aliases: dp]
filter Filter records for Alignment file [aliases: fl]
maf2sam TEST: maf2sam [aliases: m2s]
help Print this message or the help of the given subcommand (s)
Options:
-h, --help Print help (see more with '--help')
-V, --version Print version
GLOBAL:
-o, --outfile <OUTFILE> Output file ("-" for stdout) [default: -]
-r, --rewrite Bool, if rewrite output file [default: false]
-t, --threads <THREADS> Threads, default 1 [default: 1]
-v, --verbose... Logging level [-v: Info, -vv: Debug, -vvv: Trace, defalut: Warn]
其中有些参数是全局的,这些在所有子命令中都是通用的,比如指定输出的文件,指定是否覆盖已有的文件,指定线程数,指定日志等级。
同时提供了 alias 的重命名,可以不用输入完整的命令,比如 maf2paf 可以简写为 m2p, paf2maf 可以简写为 p2m 等等。当然这个功能只是让用户更加方便。
示例
格式的互相转换
这个是本工具的核心功能,用于三种格式之间的互相转换。用法简单,以 maf2paf 为例:
$ wgatools maf2paf test.maf > test.paf
$ wgatools paf2maf test.paf -t target.fa -q query.fa > test.maf
支持标准输入输出,所有可以套娃:
$ cat test.maf | wgatools maf2paf | wgatools paf2maf -t target.fa -q query.fa > test.maf
$ wgatools paf2chain test.paf | wgatools chain2maf -t target.fa -q query.fa | wgatools maf2chain | wgatools chain2paf > funny.paf
MAF 的索引和区间提取
其实 MAF 是包含了全基因组比对最原始和最全面的信息,所以我经常看 MAF 文件,有时候会有需求想看指定区间的比对情况,那么 MAF 文件一行又非常长,而且没有坐标的提示,很难找到感兴趣的区间。光是提取指定的染色体比对行,就很麻烦,要看在第几行,然后再用sed提取。所以我就想可以快速提取指定染色体和区间的比对信息,于是就开发了 MAF 的索引和提取功能。示例如下:
$ wgatools maf-index test.maf
该功能无需指定输出文件,也不会输出到标准输出,会自动产生后缀为 .index 的索引文件,我将其设计成了json格式,因为这个包含的数据其实不多,没必要用上protobuf或者bincode之类的二进制格式。
有了索引之后就可以提取区间,支持两种方式的区间:
chr:start-end形式的区间
这里我采用了正则表达式方案来匹配输入,所以start和end如果是非数字会报错。同时会检查start是否小于end.
可以用逗号分隔多个区间,比如:
$ wgatools maf-extract test.maf -r chr1:1-10,chr2:66-888,chr3:100-50,chr_no:1-10,x:y-z
这个例子中的chr3:100-50 , chr_no:1-10,x:y-z都会报错:
ERROR Parse Genome Region Error By: Region `x:y-z` is match the format of `chr:start-end`
ERROR Parse Genome Region Error By: Start `100` is larger than end `50`
bed格式文件的输入
可以在文件中指定多个区间,每行一个,格式为chr\tstart\tend,比如:
$ wgatools maf-extract test.mfa -f region.bed
这两种区间输入方式,可以同时指定,会合并在一起。
如果输入的区间有一个格式错了,就会退出程序。
如果输入的区间没有匹配,会警告未匹配的区间,并且跳过。
统计
支持 MAF/PAF 格式,可以支持是否合并同一染色体,如下:
$ wgatools stat test.maf
ref_name ref_size ref_start query_name query_size query_start aligned_size unaligned_size identity similarity matched mismatched ins_event del_event ins_size del_size inv_event inv_size inv_ins_event inv_ins_size inv_del_event inv_del_size
XXX.chr2 243675191 232285008 YYY.chr2 241186694 229666595 11007552 232667639 0.47867247 0.4921803 5269012 148688 11831 11746 5229899 5315762 1 667431.0 1115 546710 1089 274090
XXX.chr6 181357234 173309259 YYY.chr6 178141476 169794562 7338167 174019067 0.5532852 0.5652489 4060099 87792 8540 8825 3361418 3190276 0 0.0 0 0 0 0
XXX.chr8 182411202 142613895 YYY.chr8 184335743 142573702 2447590 179963612 0.42162004 0.43657106 1031953 36594 0 0 0 0 1 2241621.5 2575 967106 2532 1379043
XXX.chr9 163004744 162104206 YYY.chr9 166955280 166049855 792919 162211825 0.64267725 0.652846 509591 8063 489 484 301376 275265 0 0.0 0 0 0 0
$ wgatools stat test.maf -e
ref_name ref_size ref_start query_name query_size query_start aligned_size unaligned_size identity similarity matched mismatched ins_event del_event ins_size del_size inv_event inv_size inv_ins_event inv_ins_size inv_del_event inv_del_size
XXX.chr2 243675191 239959575 YYY.chr2 241186694 237977650 531121 0 0.45960337 0.48394057 244105 12926 0 0 0 0 1 667431.0 1115 546710 1089 274090
XXX.chr2 243675191 240631599 YYY.chr2 241186694 238865793 2853246 0 0.4797739 0.48980528 1368913 28622 2692 2662 718206 1455711 0 0.0 0 0 0 0
XXX.chr2 243675191 232285008 YYY.chr2 241186694 229666595 7623185 0 0.47958878 0.49364328 3655994 107140 9139 9084 4511693 3860051 0 0.0 0 0 0 0
XXX.chr6 181357234 173309259 YYY.chr6 178141476 169794562 7338167 0 0.5532852 0.5652489 4060099 87792 8540 8825 3361418 3190276 0 0.0 0 0 0 0
XXX.chr8 182411202 142613895 YYY.chr8 184335743 142573702 2447590 0 0.42162004 0.43657106 1031953 36594 0 0 0 0 1 2241621.5 2575 967106 2532 1379043
XXX.chr9 163004744 162104206 YYY.chr9 166955280 166049855 792919 0 0.64267725 0.652846 509591 8063 489 484 301376 275265 0 0.0 0 0 0 0
过滤
在某些比对中,我们会有contig和chromosome比对到一起的情况,通常我们并不需要这些比对,所以可以过滤掉。
$ wgatools filter test.maf -q 1000000 > filt.maf
鉴定变异
MAF 格式完整地记录了每个碱基的比对情况,所以可以用来鉴定变异,支持SNP/INS/DEL/INV这几种显式的变异类型。默认参数不输出SNP和长度小于 50 的INS和DEL,示例如下:
$ wgatools call test.maf > call.vcf
本功能只支持鉴定显式的变异,如果要鉴定DUP等其他染色体重排变异,还需要提取上下游序列来比对预测,也许之后可以加上。
终端可视化
这个更是重量级,是我开发过程中最中意的一个功能。
之前提到, 查看 MAF 文件非常麻烦,而且就算提取了指定区间,也没有坐标的指示,而且我想换一下 target 和 query 的上下位置也很麻烦,所以就直接开发了一个终端可视化的功能,可以在终端中查看 MAF 文件,使用方法如下:
$ wgatools tview test.maf
会出现一个终端可视化页面,如下:

进入该页面后,按下 ◄► 可以左右滑动,在输入参数中可以指定滑动步长,默认为 10,按下 q 可以退出。
按下 g 可以调出导航窗口,其中左侧为可选的序列名称,右侧为选中序列的可选区间,可以按下 Tab切换左右选择窗口,可以按▲▼来选择序列和区间,同时下方的输入框会随之更新,也可以回车删除和键盘输入来指定区间,检查输入是合法区间后,可以按下 Enter 可以跳转到指定的序列和区间,同时会退出导航窗口,也可以按下Esc来退出导航窗口。
开发历程
其实这个项目有了想法之后,是从 7 月初开始动手的,当时看到泛基因组圈大佬Erik实验室的一个博后用 rust 写了一个 paf 转 chain 的工具, 当时我抱着学习的心态也想复刻一下这个简单的功能,但是我在其中想用更高效更合理的方法,也算是改进了一下,实现了第一个功能。当初测试我的速度比他快十几倍,这大大增强了我的信心,原来我也不差啊😄:
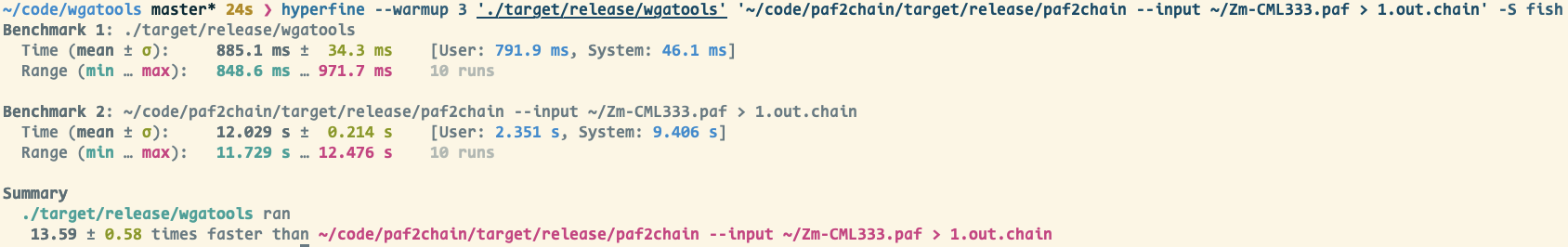
然后一不做二不休,我就把其他转换的功能也实现了,也陆陆续续加了其他功能。
最值得高兴的事情是,11 月的一个半夜,欧洲的某个师兄和我说,Erik 在泛基因组的群聊中转发了我的工具,然后我马上登了一下 Github, 发现果然也点赞了,然后圈里的挺多人也跟着点赞了,当时给我整激动了。

这一系列工具,其实没有课题支持,完全是出于公益和开源性质,同时也想提高一下自己的实践能力。
每年快到年末的时候我都会问自己,成为去年时候想成为的那个人了吗?犹记得一两年前刚接触泛基因组和图形基因组的时候,拜读了 Erik 的剑桥大学的博士毕业论文,被这个领域彻底吸引,对圈内的人物也算悉数了解,也梦想过能踏入这个圈子。从一开始给 vg 系列软件提 issue 和想法,也帮助别人解决问题,到后来自己写的软件能被 Erik 转发,并由此参与到项目中。回到开头的那个提问,我觉得我做到了。
开发经验
开发过程中并非顺风顺水,遇到过不少问题,也提升了不少技能,这里简单记录一下。
组合而非继承
在本例中,不管哪种格式,其本质都是一样的,无非是部分属性的有无。比如几种格式都可以总结为target和query的比对信息,包括名称,长度,起始位置,方向,等等。如果用 rust 来实现,就可以将这类属性抽象为一个trait,然后每个格式都实现这个trait. 当需要对某个比对进行操作时,参数可以是实现了这个trait的任意格式,这样就可以实现对不同格式的操作,而不用写多个函数。这就是组合而非继承的思想。
项目的结构
本例中,采取了库和可执行文件并存的方式,因此要兼顾库的可复用性,其中我对多种文件格式的读取和迭代器进行了统一来保证库使用的便利性,同时也提供了多种接口。
对于可执行文件,重点在于参数的解析和调用,需要封装子命令所需要的函数。
错误的处理
其实库和可执行文件的错误处理方式并不一样,但好在可以传递。
通常来讲,库中的接口,负责返回错误,也就是Result<T, E>, 最好不要有unwrap(), 因为这样会导致调用者只能看到panic的信息,无法处理。
而对于 CLI 的处理,拿到封装函数的结果后,可以用一个最简单的match来处理,本例中的真实情况如下:
fn main() {
match main_entry() {
Ok(_) => {}
Err(e) => {
error!("{}", e);
std::process::exit(1);
}
}
}
这里我将 CLI 可能得到的错误都集中到main_entry()中,如果有错误,就会打印具体是哪个错误,这样就很好的做到了错误的管理和显示。
当然这里还有很多值得分享和记录的点,开个坑,记录在这里
