隐式二叉搜索树 (implicit BST)
背景
Erik: https://github.com/pangenome/impg
lh3: https://github.com/lh3/bedtk
两位重量级都提到了这个概念,必须要学习一下。而且听起来也很高端的样子。
定义
隐式数据结构
维基百科定义:
In computer science, an implicit data structure or space-efficient data structure is a data structure that stores very little information other than the main or required data: a data structure that requires low overhead. They are called "implicit" because the position of the elements carries meaning and relationship between elements; this is contrasted with the use of pointers to give an explicit relationship between elements.
TL;DR: 隐式数据结构是一种空间高效,低开销的数据结构,为什么叫隐式,因为元素的位置本身就包含了元素之间的关系,而不是通过指针来明确元素之间的关系。
乍一听,是乍一听的感觉。
二叉搜索树
这个老面孔了,简单的说:
二叉搜索树(binary search tree)满足以下条件。
- 对于根节点,左子树中所有节点的值 < 根节点的值 < 右子树中所有节点的值。
- 任意节点的左、右子树也是二叉搜索树,即同样满足条件 1。
如下图所示,这样的特性是为了检索方便。
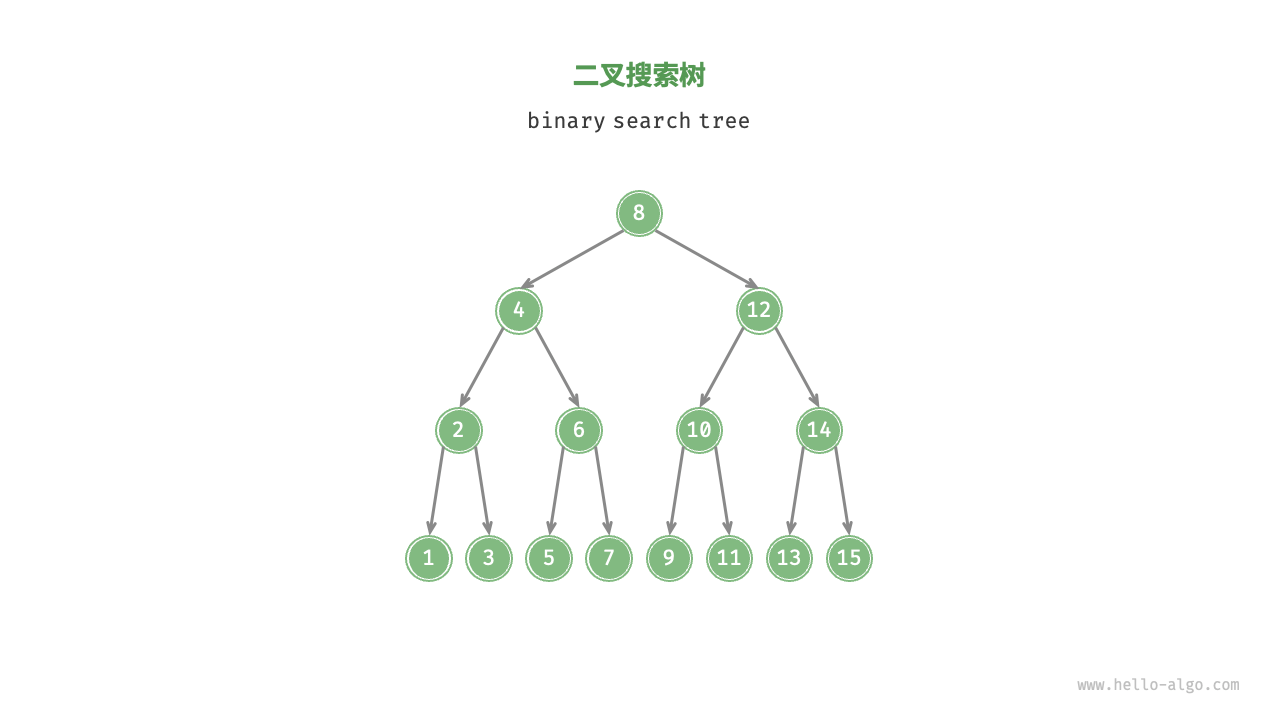
具体的检索过程是,假定要查找的值为 ,从根节点开始,如果 小于当前节点的值,则在左子树中查找,否则在右子树中查找,直到找到为止。
隐式二叉搜索树
Q: 我们真的需要用树结构来存储一个 BST 吗?
A: 不用,可以用隐式数据结构来代替。
具体而言,我们可以用一个排序好的数组来代替二叉搜索树,这样就不需要额外的指针来存储左右子树的信息了。 其中数组的下标就是节点的位置,节点的值就是数组的值。如下图所示:
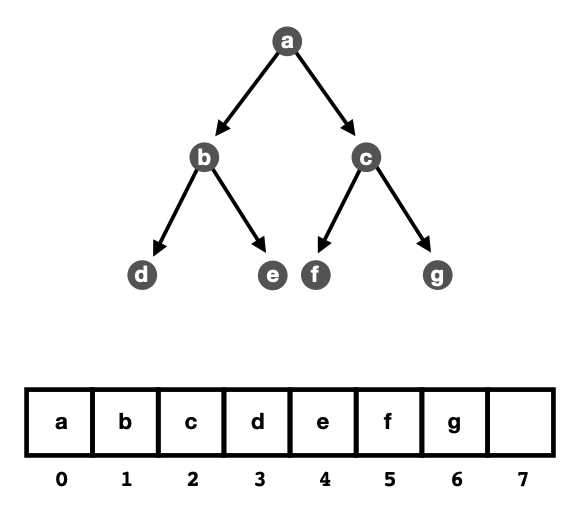
左右子节点,有不同的索引下标,具体如图所示:
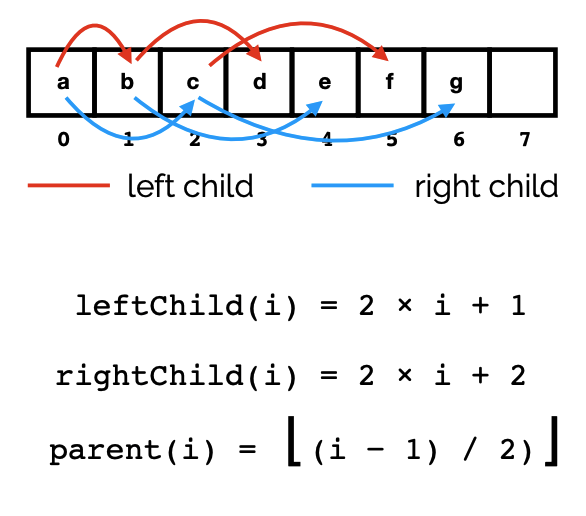
lh3 那篇 bedtk 的文章也阐述地很清楚:
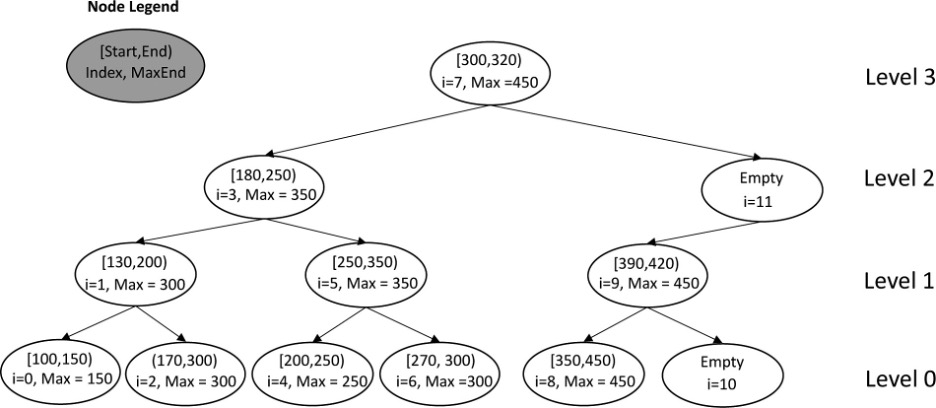
在这个例子中,BST 总共有 3 层,第一个根节点的索引就是,左子节点的索引是,右子节点的索引是。以此类推。
优势
- 空间高效: 数组实现,不需要存储指针或引用。
- 缓存: 由于数组是连续存储的,可以利用 CPU 缓存的特性,提高检索效率。
- 简单: 结构简单,实现容易
劣势
- 灵活性差: 由于数组是连续存储的,插入删除操作需要移动大量元素,代价太大。
- 场景有限: 适用于静态数据集,不适用于动态数据场景。
实现
这里仅实现了一个简单的隐式二叉搜索树的检索过程,省略了隐式数组的生成,具体代码如下:
struct ImplicitBST {
data: Vec<i32>, // 数组存储 BST
}
impl ImplicitBST {
pub fn new(data: Vec<i32>) -> Self {
ImplicitBST { data }
}
pub fn search(&self, value: i32) -> Option<usize> {
let mut idx = 0;
while idx < self.data.len() {
if self.data[idx] == value {
return Some(idx);
} else if self.data[idx] > value {
idx = 2 * idx + 1; // 左子节点
} else {
idx = 2 * idx + 2; // 右子节点
}
}
None
}
}
fn main() {
let ibst = ImplicitBST::new(vec![10, 5, 15, 3, 7, 12, 18]); // 已经排序好的数组,第一个节点为根节点,左节点索引为 2*i+1, 右节点索引为 2*i+2
// 因此这个例子的树结构为:
// 10
// / \
// 5 15
// / \ / \
// 3 7 12 18
println!("Search for 7: {:?}", ibst.search(7)); // Output: Some(4)
println!("Search for 3: {:?}", ibst.search(3)); // Output: Some(3)
println!("Search for 20: {:?}", ibst.search(20)); // Output: None
}
impg 的原理
据我所知,impg 设计的初衷是不想构建整个完整的 graph, 因为由于种种原因,完整的 pan-graph 会变得很复杂。一个解决方法是获得指定区间的子图,也就是指定区间的比对信息。
具体而言,构建 pan-graph 的前期,会对每个基因组进行两两之间的 all-vs-all 比对,那么每个基因的比对信息,其实就是一组区间。
用户指定一个基因组的区间,impg 就会去找每个 alignments 上有交集的区间,对于找到的交集区间,再重复上述过程,直到找到所有的区间。
这样找到的比对信息,是一个找 传递闭包的过程,也就是说,如果 A 和 B 有比对信息,B 和 C 有比对信息,那么 A 和 C 也有比对信息。
这样,就完成了一个 sub-graph 的检索,这样的操作也会 more flexible, parallelizable, and scalable.
